用 Python 进行贝叶斯模型建模(2),python贝叶斯,未经许可,禁止转载!英文
用 Python 进行贝叶斯模型建模(2),python贝叶斯,未经许可,禁止转载!英文
本文由 编橙之家 - JLee 翻译,sunshinebuel 校稿。未经许可,禁止转载!英文出处:markdregan。欢迎加入翻译组。
本系列:
- 《第 0 节:导论》
- 《第 1 节:估计模型参数》
- 《第 2 节:模型检验》
- 《第 3 节:分层模型》
- 《第 4 节:贝叶斯回归》
- 待续
第2节:模型检验
在这一节中,我们将用到两种技术,旨在回答:
- 模型和估计的参数是否对潜在数据有很好的模拟?
- 给定两个独立的模型,哪个对潜在数据模拟的更好?
模型检验I:后验估计检验
一种检验模型拟合的方法是后验估计检验。这种方法很直观,回顾上节中,我们通过收集 200,000 个 μ 的后验分布样本来对泊松分布的参数 μ进行估计,每个样本都被认为是可信的参数值。
后验预测检验需要从预测模型中产生新的数据。具体来说就是,我们已经估计了 200,000 个可信的泊松分布参数值μ,这意味着我们可以根据这些值建立 200,000 个泊松分布,然后从这些分布中随机采样。用公式表示为:

理论上,如果模型对潜在数据拟合的很好,那么产生的数据应该和原始观测数据近似。PyMC 提供一种方便的方式来从拟合模型中采样。你可能已经注意到了上面模型应用中新的一行代码:
Python
y_pred = pm.Poisson('y_pred', mu=mu)
这几乎和 y_est 一样,只不过没给观测数据赋值。PyMC 把它当做随机节点(和观测节点相反),当 MCMC 采样器运行时,它也从 y_est 中采集数据。
然后画出y_pred并和观测数据y_est比较。
Python
import json
import matplotlib.pyplot as plt
import numpy as np
import pandas as pd
import pymc3 as pm
import scipy
import scipy.stats as stats
import statsmodels.api as sm
import theano.tensor as tt
from IPython.display import Image
%matplotlib inline
plt.style.use('bmh')
colors = ['#348ABD', '#A60628', '#7A68A6', '#467821', '#D55E00',
'#CC79A7', '#56B4E9', '#009E73', '#F0E442', '#0072B2']
messages = pd.read_csv('data/hangout_chat_data.csv')
Python
Applied interval-transform to mu and added transformed mu_interval to model. [-----------------100%-----------------] 200000 of 200000 complete in 63.5 secPython
x_lim = 60
burnin = 50000
y_pred = trace[burnin:].get_values('y_pred')
mu_mean = trace[burnin:].get_values('mu').mean()
fig = plt.figure(figsize=(10,6))
fig.add_subplot(211)
_ = plt.hist(y_pred, range=[0, x_lim], bins=x_lim, histtype='stepfilled', color=colors[1])
_ = plt.xlim(1, x_lim)
_ = plt.ylabel('Frequency')
_ = plt.title('Posterior predictive distribution')
fig.add_subplot(212)
_ = plt.hist(messages['time_delay_seconds'].values, range=[0, x_lim], bins=x_lim, histtype='stepfilled')
_ = plt.xlabel('Response time in seconds')
_ = plt.ylabel('Frequency')
_ = plt.title('Distribution of observed data')
plt.tight_layout()
选择正确的分布
我对上面的结果不是很满意。理想情况下,我希望后验预测分布和观测数据的分布一定程度上近似。直观上,如果我们正确估计了模型参数,那么我们应该可以从模型中采样得到类似的数据。结果很明显不是这样。
可能泊松分布不适合拟合这些数据。一种可选模型是负二项分布,特点和泊松分布很像,只是有两个参数(μ 和 α),使得方差和均值独立。回顾泊松分布只有一个参数 μ,既是均值,又是方差。
Python
fig = plt.figure(figsize=(10,5))
fig.add_subplot(211)
x_lim = 70
mu = [15, 40]
for i in np.arange(x_lim):
plt.bar(i, stats.poisson.pmf(mu[0], i), color=colors[3])
plt.bar(i, stats.poisson.pmf(mu[1], i), color=colors[4])
_ = plt.xlim(1, x_lim)
_ = plt.xlabel('Response time in seconds')
_ = plt.ylabel('Probability mass')
_ = plt.title('Poisson distribution')
_ = plt.legend(['$lambda$ = %s' % mu[0],
'$lambda$ = %s' % mu[1]])
# Scipy takes parameters n & p, not mu & alpha
def get_n(mu, alpha):
return 1. / alpha * mu
def get_p(mu, alpha):
return get_n(mu, alpha) / (get_n(mu, alpha) + mu)
fig.add_subplot(212)
a = [2, 4]
for i in np.arange(x_lim):
plt.bar(i, stats.nbinom.pmf(i, n=get_n(mu[0], a[0]), p=get_p(mu[0], a[0])), color=colors[3])
plt.bar(i, stats.nbinom.pmf(i, n=get_n(mu[1], a[1]), p=get_p(mu[1], a[1])), color=colors[4])
_ = plt.xlabel('Response time in seconds')
_ = plt.ylabel('Probability mass')
_ = plt.title('Negative Binomial distribution')
_ = plt.legend(['$mu = %s, / beta = %s$' % (mu[0], a[0]),
'$mu = %s, / beta = %s$' % (mu[1], a[1])])
plt.tight_layout()
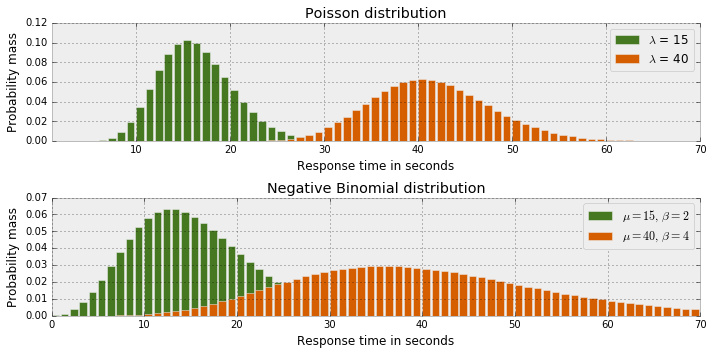
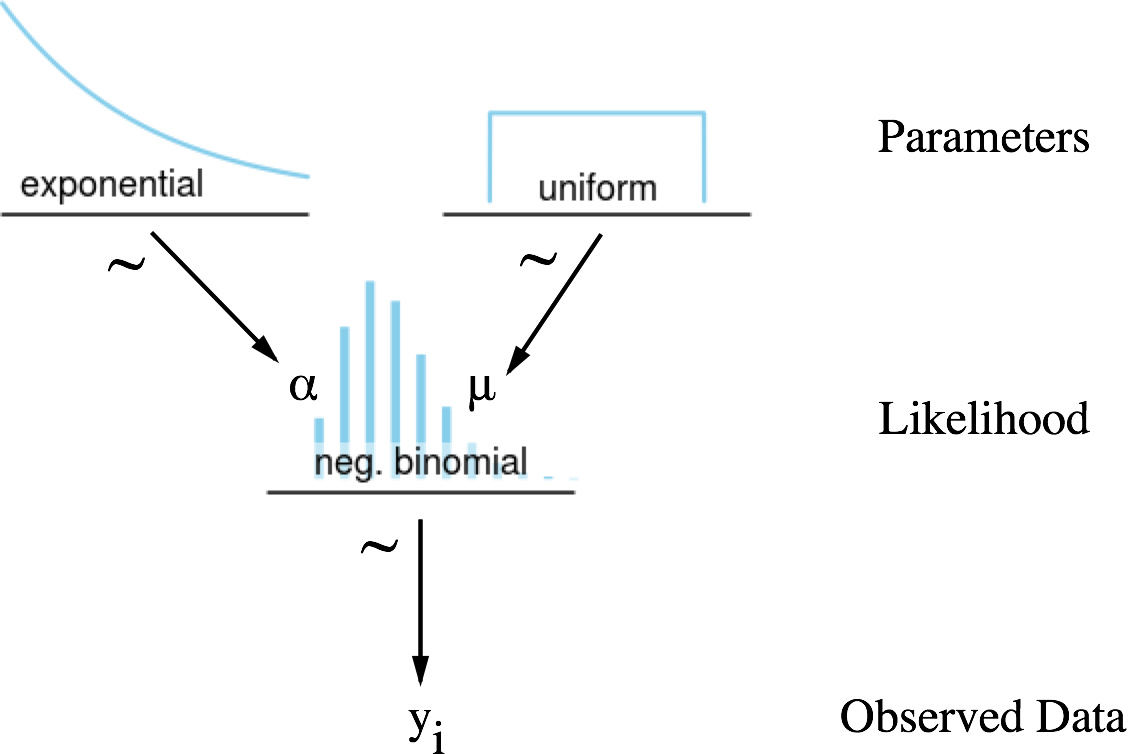
使用之前相同的数据集,继续对负二项分布的参数进行估计。同样地,使用均匀分布来估计 μ 和 α。模型可以表示为:



代码:
Python
Image('graphics/Neg Binomial Dag.png', width=400)
with pm.Model() as model:
alpha = pm.Exponential('alpha', lam=.2)
mu = pm.Uniform('mu', lower=0, upper=100)
y_pred = pm.NegativeBinomial('y_pred', mu=mu, alpha=alpha)
y_est = pm.NegativeBinomial('y_est', mu=mu, alpha=alpha, observed=messages['time_delay_seconds'].values)
start = pm.find_MAP()
step = pm.Metropolis()
trace = pm.sample(200000, step, start=start, progressbar=True)
Applied log-transform to alpha and added transformed alpha_log to model. Applied interval-transform to mu and added transformed mu_interval to model. [-----------------100%-----------------] 200000 of 200000 complete in 114.8 secPython
_ = pm.traceplot(trace[burnin:], varnames=['alpha', 'mu'])
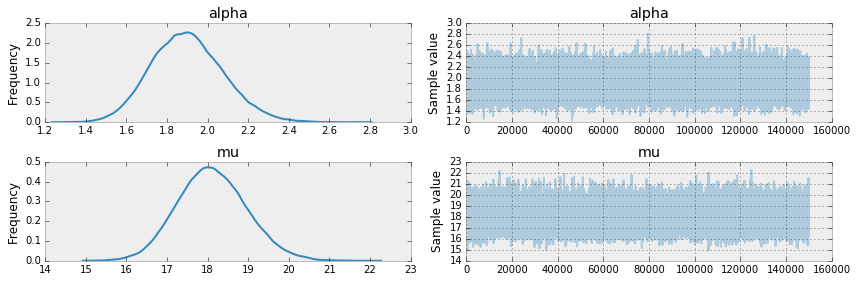
我们看到上面的模型在平均回复时间 μ 附近有更大的不确定度:
- 泊松分布:17.5 到 18.5
- 负二项分布:16 到 21
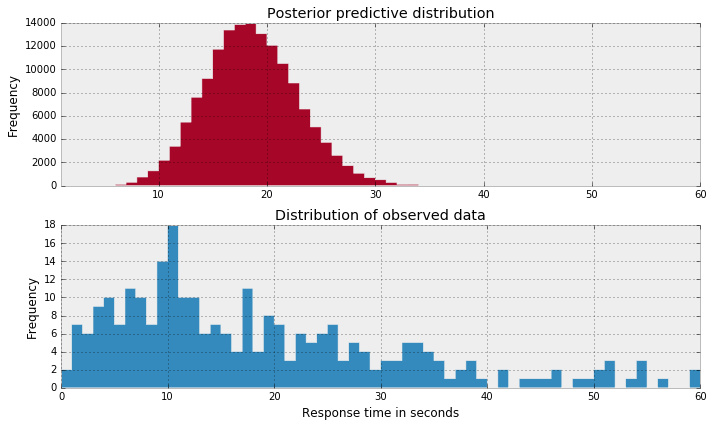
同时,负二项分布模型有一个 1.4 到 2.4 的 α 参数,增加了估计参数 μ 的方差。让我们看看后验预测分布,是否和观测数据更加近似。
Python
x_lim = 60
y_pred = trace[burnin:].get_values('y_pred')
fig = plt.figure(figsize=(10,6))
fig.add_subplot(211)
fig.add_subplot(211)
_ = plt.hist(y_pred, range=[0, x_lim], bins=x_lim, histtype='stepfilled', color=colors[1])
_ = plt.xlim(1, x_lim)
_ = plt.ylabel('Frequency')
_ = plt.title('Posterior predictive distribution')
fig.add_subplot(212)
_ = plt.hist(messages['time_delay_seconds'].values, range=[0, x_lim], bins=x_lim, histtype='stepfilled')
_ = plt.xlabel('Response time in seconds')
_ = plt.ylabel('Frequency')
_ = plt.title('Distribution of observed data')
plt.tight_layout()
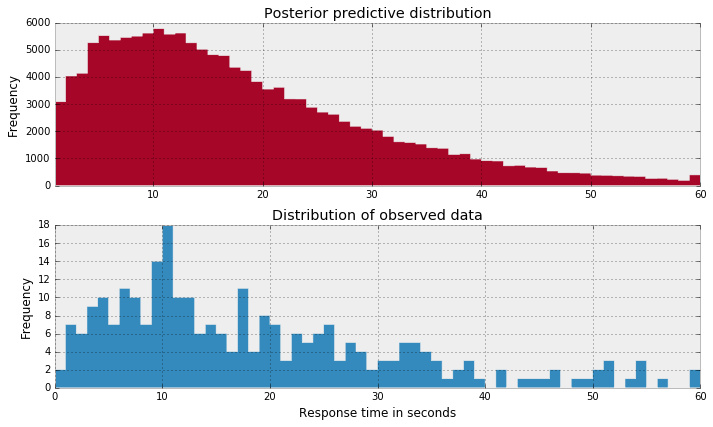
是的,和另一个相比,这两个分布看起来更近似。根据后验预测检验,负二项分布模型对潜在数据模拟更好。
如果你怀疑这种模型检验方法的严密性,贝叶斯方法还有一种更严密的分析方法。
模型检验II:贝叶斯因子
另一种模型检验的方法是计算贝叶斯因子,这种分析方法旨在两个模型间的相互比较。
贝叶斯因子通常难以计算,因为需要整合全联合概率分布。在低维空间中,整合是可能的,但是一旦你稍微增加建模的维度,整合全联合概率分布的计算会变得昂贵且费时。
有一种可选的近似方法来计算贝叶斯因子。它需要把两个模型进行比较,把它们融合成一个带有一个模型指数(τ)的分层模型.这个指数在MCMC 过程中,会根据模型的可信度在两个模型中切换,这样,这个指数的轨迹就充分显示模型 M1 相对模型 M2 的可信度。
Python
Image('graphics/Bayes Factor DAG.png', width=540)
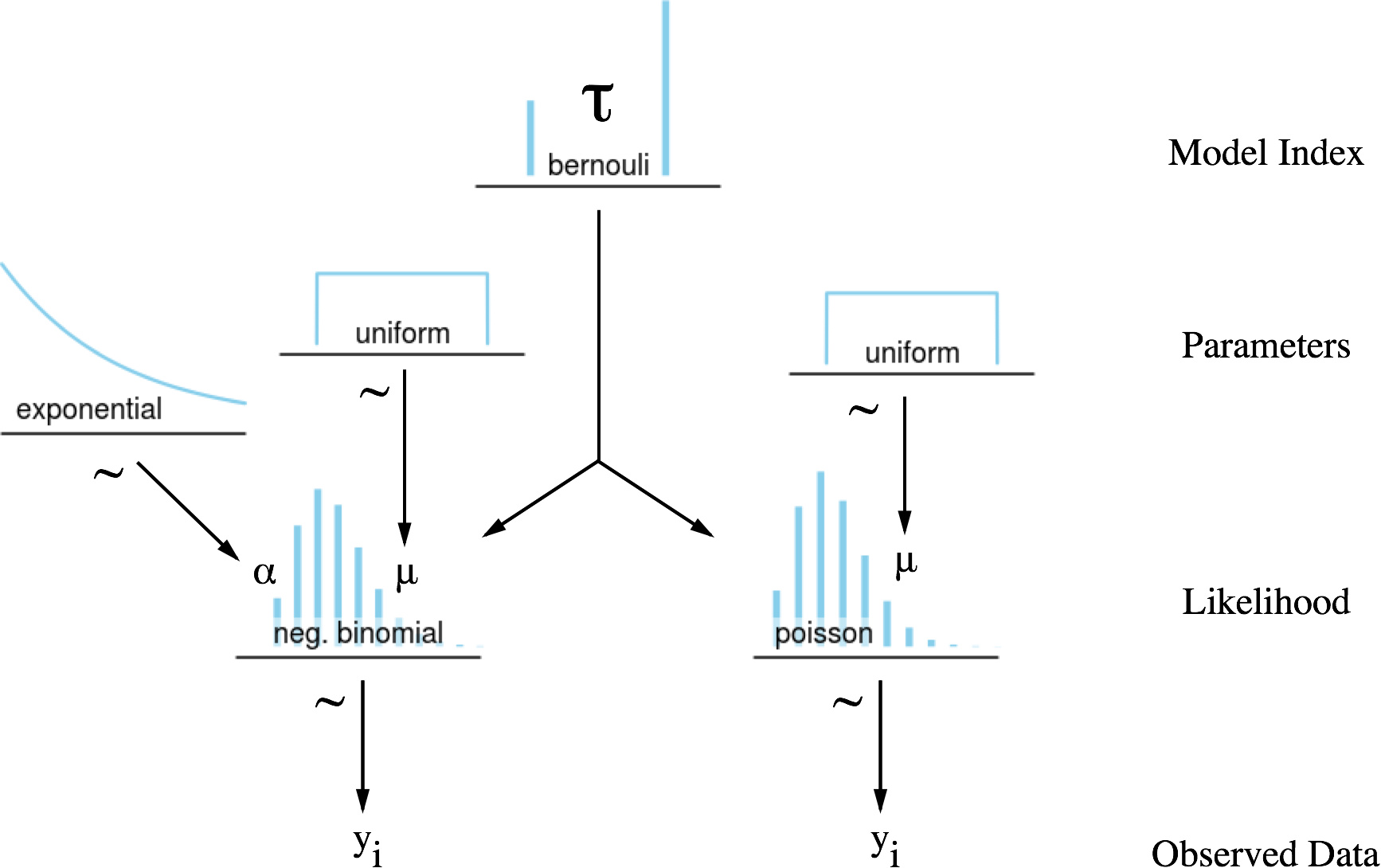
with pm.Model() as model:
# Index to true model
prior_model_prob = 0.5
#tau = pm.DiscreteUniform('tau', lower=0, upper=1)
tau = pm.Bernoulli('tau', prior_model_prob)
# Poisson parameters
mu_p = pm.Uniform('mu_p', 0, 60)
# Negative Binomial parameters
alpha = pm.Exponential('alpha', lam=0.2)
mu_nb = pm.Uniform('mu_nb', lower=0, upper=60)
y_like = pm.DensityDist('y_like',
lambda value: pm.switch(tau,
pm.Poisson.dist(mu_p).logp(value),
pm.NegativeBinomial.dist(mu_nb, alpha).logp(value)
),
observed=messages['time_delay_seconds'].values)
start = pm.find_MAP()
step1 = pm.Metropolis([mu_p, alpha, mu_nb])
step2 = pm.ElemwiseCategoricalStep(vars=[tau], values=[0,1])
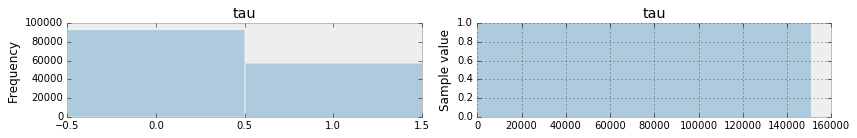
trace = pm.sample(200000, step=[step1, step2], start=start)
_ = pm.traceplot(trace[burnin:], varnames=['tau'])
Python
Applied interval-transform to mu_p and added transformed mu_p_interval to model. Applied log-transform to alpha and added transformed alpha_log to model. Applied interval-transform to mu_nb and added transformed mu_nb_interval to model. [-----------------100%-----------------] 200000 of 200000 complete in 314.6 sec
根据下面的公式计算上面两个模型的贝叶斯因子:


在上例中,我们没有使用先验概率,因此贝叶斯因子只是模型似然度的商。如果你发现你的 MCMC 样本不在两个模型间游走,你可以引入先验概率来帮助你更深入了解两个模型。
Python
# Compute the Bayes factor
prob_pois = trace[burnin:]['tau'].mean()
prob_nb = 1 - prob_pois
BF = (prob_nb/prob_pois)*(prior_model_prob/(1-prior_model_prob))
print("Bayes Factor: %s" % BF)
Python
Bayes Factor: 1.60114103387
贝叶斯因子大于 1 说明 M1(负二项分布)比 M2(泊松分布)更适合拟合数据。Jeffrey的贝叶斯因子优势判别准则把贝叶斯因子为 1.60 标识为 M1 对 M2 有弱优势。结合后验预测检验和贝叶斯因子,可得出结论:负二项分布模型更优。
| Bayes Factor | Interpretation |
|---|---|
| BF(M1,M2) < 1/10 | Strong evidence for M2 对 M2 有强优势 |
| 1/10 < BF(M1,M2)< 1/3 | Moderate evidence for M2 对 M2 有中等优势 |
| 1/3 < BF(M1,M2) < 1 | Weak evidence for M2 对 M2 有弱优势 |
| 1 < BF(M1,M2) < 3 | Weak evidence for M1 对 M1 有弱优势 |
| 3 < BF(M1,M2) < 10 | Moderate evidence for M1 对 M1 有中等优势 |
| BF(M1,M2) > 10 | Strong evidence for M1 对 M1 有强优势 |
打赏支持我翻译更多好文章,谢谢!
打赏译者
打赏支持我翻译更多好文章,谢谢!
任选一种支付方式


评论关闭